| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | ||||
| 4 | 5 | 6 | 7 | 8 | 9 | 10 |
| 11 | 12 | 13 | 14 | 15 | 16 | 17 |
| 18 | 19 | 20 | 21 | 22 | 23 | 24 |
| 25 | 26 | 27 | 28 | 29 | 30 | 31 |
- 안드로이드스튜디오
- 컴퓨터비전
- level4
- Matrix Factorization
- 다시풀기
- level3
- LEVEL2
- WebOS
- 내휴학생활중의아주큰일
- SQL
- 어렵다
- 휴학
- 파이썬
- D3
- MSBuild
- 대학원
- py
- level1
- git
- BFS
- 대학원일기
- androidstudio
- build
- Github
- SWEA
- java
- 자바
- 프로그래머스
- Python
- 컨트리뷰톤
- Today
- Total
bit가 눈 앞에서 왔다갔다
나의 Matrix Factorization 이야기 (1) 본문
논문 하나를 봤는데, MF의 MAE, RMSE, PCC가 너무 높게 나왔다.
교수님께서 공부할 겸, 얼마나 이상한 수치인지를 확인해보라고 말씀하셔서 넹~~ 하고 MF를 엄청 많이 다뤄본 박사과정 선배님한테 조언 구하면서 코딩했다.
내가 만든 모델은 그렇게 엄청난 값을 뱉어냈고, 선배들에게 광대역 어그로를 끈 뒤, 며칠 뒤 관련 분야 논문이 나오는 언니가 도와주셔서(밥 먹다가 0.9 나오는 이야기 듣고 충격 먹으셨다고..) 이상한 상태를 알게되었다.
(저 신입생이 MF로 PCC 0.9를 찍었대!! 웅성웅성) (웅성웅성) 소문난 어그로 맛집...ㅠ
제대로 하려면 코딩을 처음부터 다시 짜야하는데, 이건 어쨌든 여기까지의 시행착오, 공부를 정리한 글이다.
1. MF 개념 실수
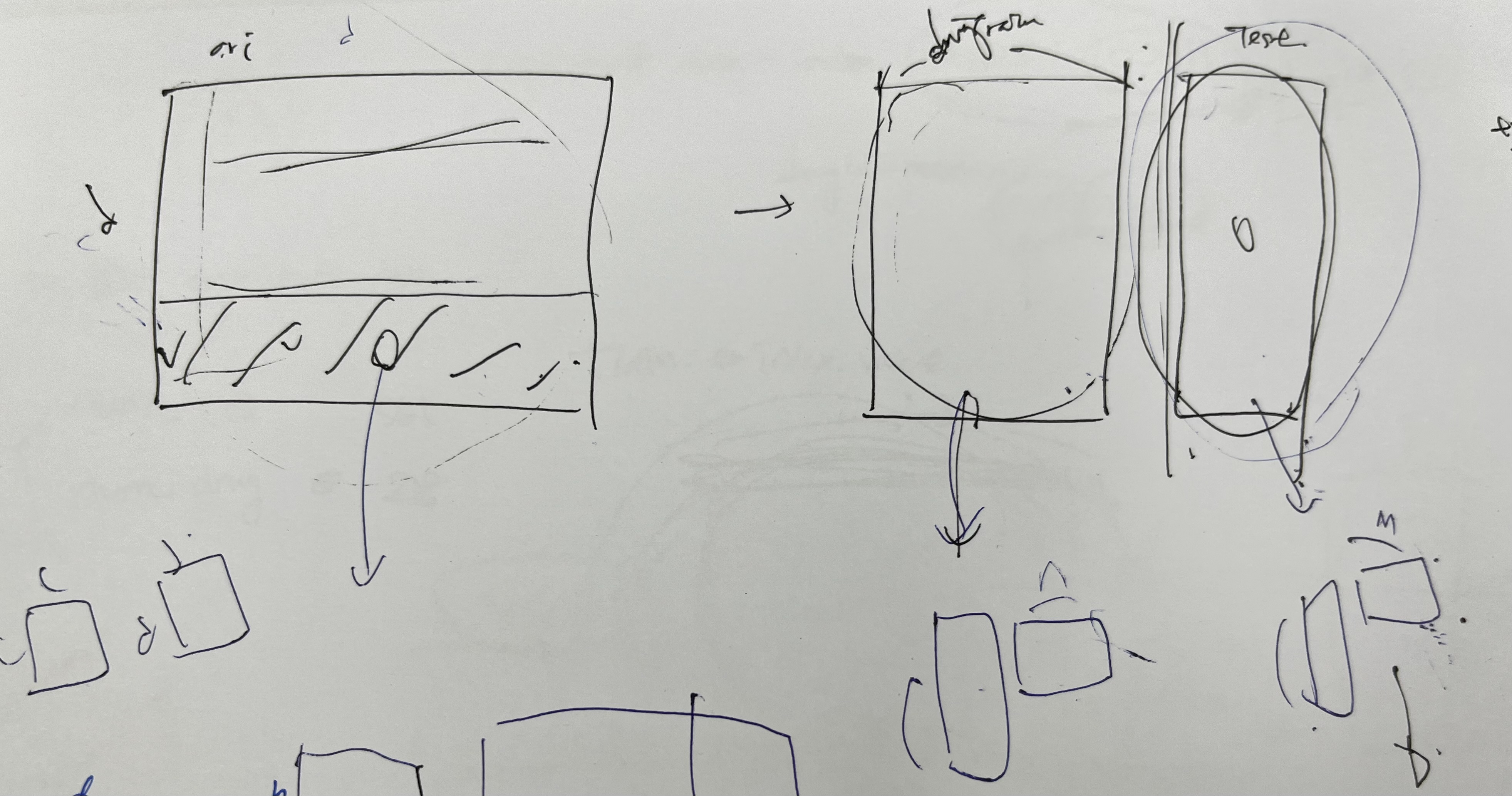
나는 이렇게 했다고 당당하게 선배님한테 설명했는데, (train, test를 분리해서 train으로 학습시키고 test로 모델 평가를 함) 이렇게 하면 안된다.
행렬곱이 일어나면서 원본 matrix의 원소 위치의 하나하나의 값을 채우는 건데, 저렇게 분리해버리면 dimension이 바뀌어버린다.
어쨌든 저런 잘못된 식으로 코딩을 했는데, 그마저도 제대로 안됐으니 잘 안된거 하나씩 살펴본다.
2. 코드 (틀림)
class MatrixFactorization(nn.Module):
def __init__(self, n_cells, n_drugs, emb_size):
super(MatrixFactorization, self).__init__()
self.cell_emb = nn.Embedding(n_cells, emb_size)
self.drug_emb = nn.Embedding(n_drugs, emb_size)
def forward(self, cell_idx, drug_idx):
cell_embedding = self.cell_emb(cell_idx)
drug_embedding = self.drug_emb(drug_idx)
return (cell_embedding * drug_embedding).sum(1)
def trainMF(matrix, test_set, target, emb_size, learning_rate, num_epochs):
device = 'cpu'
# user, item 인덱스 저장
train_index_list = [i.split("-")[1] for i in train.index]
cells = np.array(list(map(int, train_index_list)))
drugs = matrix.columns.values
n_cells, n_drugs = matrix.shape # 유저, 아이템 총 개수 저장
model = MatrixFactorization(n_cells, n_drugs, emb_size).to(device)
optimizer = torch.optim.Adam(model.parameters(), lr=learning_rate)
criterion = torch.nn.MSELoss()
for epoch in range(num_epochs):
model.train()
optimizer.zero_grad()
pred = model(torch.from_numpy(cells).to(device), torch.from_numpy(drugs).to(device))
loss = criterion(pred, torch.from_numpy(matrix[cells, drugs]).float().to(device))
loss.backward()
optimizer.step()
model.eval()
with torch.no_grad(): # gradient 연산을 끔
test_pred = model(torch.from_numpy(test_set.rows).to(device), torch.from_numpy(test_set.columns).to(device))
pcc = np.corrcoef(test_pred.cpu().detach().numpy(), target)[0, 1]
print(f"Epoch {epoch + 1}, Loss: {loss.item():.4f}, PCC: {pcc:.4f}")
return model
1) Index out of range -> embedding 문제!
main에서 matrix를 넣었을 때 index out of range라는 오류가 떴었다.
처음엔 단순히 내가 matrix index를 잘못 잡아줬나? 싶었지만 그런 문제가 아니었다.
train_index_list = [i.split("-")[1] for i in train.index]
cells = np.array(list(map(int, train_index_list)))
drugs = matrix.columns.values
저런 식으로 코드를 처음 짰었는데, 그 이유는 matrix의 인덱스가 GDSC-1001 이런식이었고, 넘파이가 지원하는 형식이 아니라길래 1001만 추출해서 np.array로 만들어주는 식으로 코딩했었다.
np.array인 train_index_list에 [1001, 1002, 1003...] 이런식으로 데이터가 들어가 있는 상황.
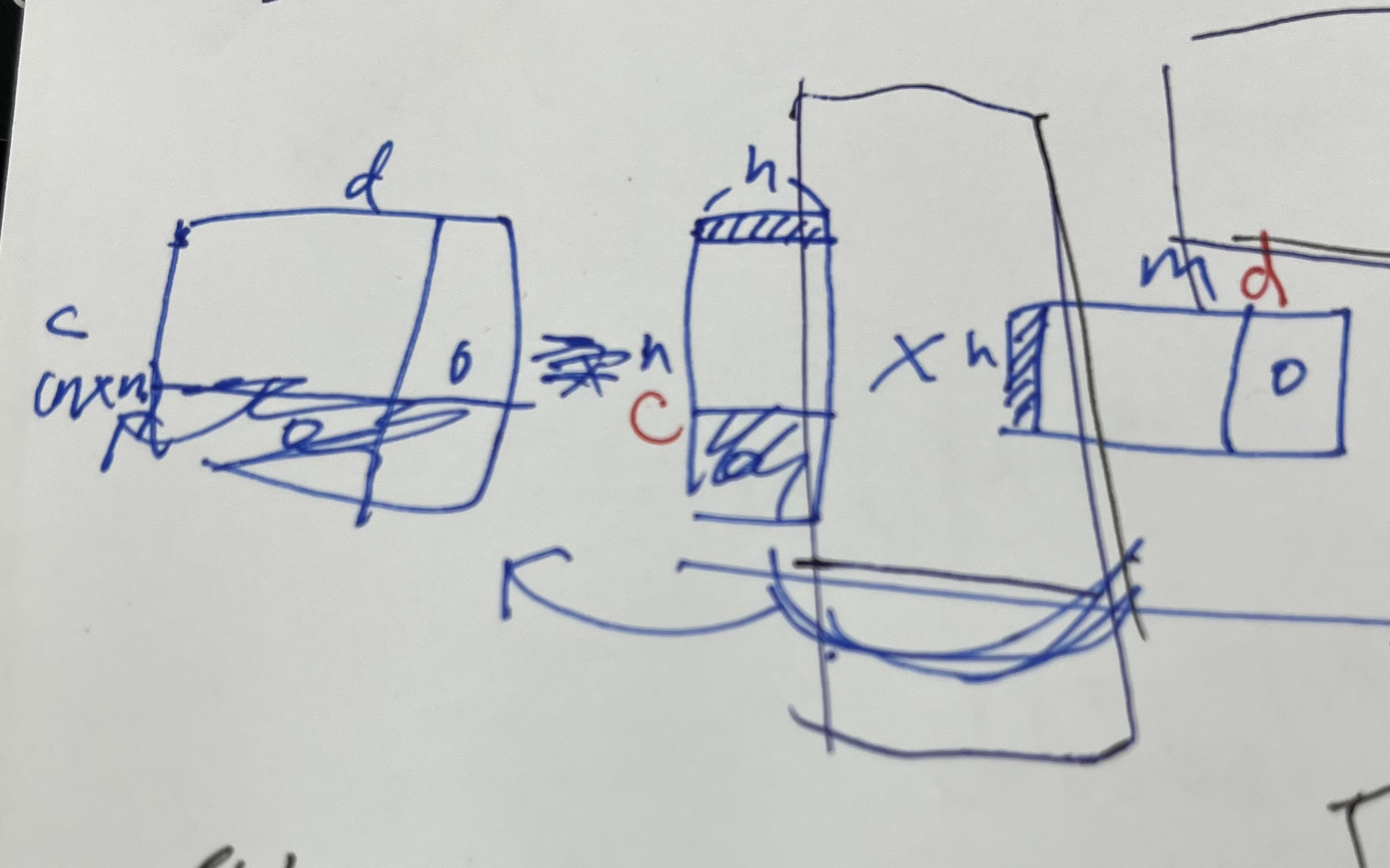
그런데 임베딩은,
1xh의 array의 원소값이 42라면 매트릭스의 42번 인덱스에서의 값을 찾아 그것과 계산하도록 해준다고 한다.
그림 설명대로라면 (b,h)일때 nxd 매트릭스에서 b와 일치하는 부분만 계산하는 식?
아직 임베딩에 대해 자세히 공부하진 않았지만 일단 그런 식으로 이해했다.
(틀리면 제발 알려주세요ㅠ)
그런데 내 train_index_list는 matrix dimension보다 더 큰 값을 가지고 있을테니까
pred = model(torch.from_numpy(cells).to(device), torch.from_numpy(drugs).to(device))
로 넘어가고
def forward(self, cell_idx, drug_idx):
cell_embedding = self.cell_emb(cell_idx)
drug_embedding = self.drug_emb(drug_idx)
return (cell_embedding * drug_embedding).sum(1)
에서 임베딩이 처리가 될때 기존에 만들어진 임베딩과 차이가 있으니까 오류를 일으킨 것이다.
2) matrix 넘기기
loss = criterion(pred, torch.from_numpy(matrix[cells, drugs]).float().to(device))
test_pred = model(torch.from_numpy(test_set.rows).to(device), torch.from_numpy(test_set.columns).to(device))
그 다음은 여기서 에러가 났던거 같은데 아마 index와 columns 이름들을 넘겨주고 싶었던것 같다. 그냥 reshape해서 넘겨주면 됐었는데 코딩하다보니 넘파이, 데이터프레임 이런 것들이 꼬여서 그랬던 것 같음.
윗줄 matrix도 그냥 넘겨주면 되는데 왜 cell, drugs라고 해놨을까..? 확인해보니까 gpt가 그랬다.
matrix 중 cells, drugs에 해당하는 부분만 추출했다고 한다. 그건 제대로된 MF라면 맞겠지만, 지금 나처럼 이상하게 해둔 상태에선 맞지 않는 방법이라 틀렸다.
3)
cells = np.arange(matrix.shape[0])
drugs = np.arange(matrix.shape[1])
이렇게 해도 되는거라 다시 제대로 바꿔줬다.
마지막 줄도 이렇게 바꿔주었다. matrix factorization은 matrix 하나를 전치해서 처리해야한다.
기존에는 return (cell_embedding * drug_embedding).sum(1) 였는데, 동기랑 같이 '도대체 .sum(1)은 왜 붙이는거야..?' 하면서 이렇게 바꿔줘버렸다.

4) PCC - 잘못 건드렸다가 어마어마한 일이 생겼다. 이거 자체가 크나큰 사고였다.
pcc = np.corrcoef(test_pred.cpu().detach().numpy().flatten(), matrix.flatten())[0, 1]
를 pcc = np.corrcoef(test_pred.cpu().detach().numpy(), matrix)[0, 1] 로 바꿔버렸다. (동기가 바꿨지만 틀린지 모르는 나도 문제 있으므로 내 잘못이다.)
PCC를 flatten해서 계산하지도 않고, [0,1]로 지정해버리는.. 어마어마한 사고를 쳤다.
나중에 선배님이 코드 보다가 잡아내심...
어쨌든 이렇게 코드를 고쳤다.
class MatrixFactorization(nn.Module):
def __init__(self, n_cells, n_drugs, emb_size):
super(MatrixFactorization, self).__init__()
self.cell_emb = nn.Embedding(n_cells, emb_size)
self.drug_emb = nn.Embedding(n_drugs, emb_size)
def forward(self, cell_idx, drug_idx):
cell_embedding = self.cell_emb(cell_idx)
drug_embedding = self.drug_emb(drug_idx)
return torch.matmul(cell_embedding, drug_embedding.T)
def trainMF(matrix, test, target, emb_size, learning_rate, num_epochs):
device = torch.device('cuda' if torch.cuda.is_available() else 'cpu')
# user, item 인덱스 저장
cells = np.arange(matrix.shape[0])
drugs = np.arange(matrix.shape[1])
n_cells, n_drugs = matrix.shape
model = MatrixFactorization(n_cells, n_drugs, emb_size).to(device)
optimizer = torch.optim.Adam(model.parameters(), lr=learning_rate)
criterion = torch.nn.MSELoss()
for epoch in range(num_epochs):
model.train()
optimizer.zero_grad()
pred = model(torch.from_numpy(cells).to(device), torch.from_numpy(drugs).to(device))
loss = criterion(pred, torch.from_numpy(np.array(matrix)).float().to(device)) ######
loss.backward()
optimizer.step()
model.eval()
with torch.no_grad(): # gradient 연산을 끔
test_pred = model(torch.from_numpy(np.arange(test.shape[0])).to(device), torch.from_numpy(np.arange(test.shape[1])).to(device))
mae = mean_absolute_error(target, test_pred.cpu())
mse = mean_squared_error(target, test_pred.cpu())
# rmse = np.sqrt(mse)
pcc = np.corrcoef(test_pred.cpu().detach().numpy(), target)[0, 1]
print(f"Epoch {epoch + 1}, Loss: {loss.item():.4f}, MAE: {mae:.4f}, RMSE: {mse:.4f}, PCC: {pcc:.4f}")
return model, mae, mse, pcc
그리고 모델을 돌린 결과는 아래와 같았다.
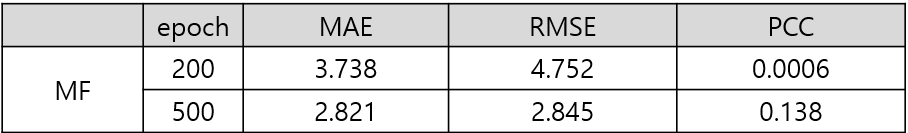
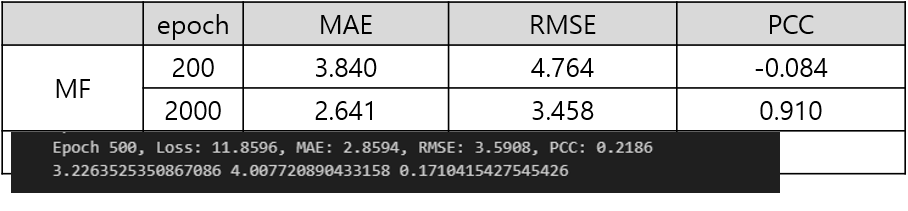
위에는 non-overlapping cell lines, non-overlapping drugs인데 drug가 0.910이라는 어마어마한 성능을 냈고, 오늘 미팅때 교수님이 보시더니 ??이거 컬럼별로 correlation 찍어오세요. 하셨다.
그리고 박사과정 선배님이 결과를 보고 충격 받으심.
'연구' 카테고리의 다른 글
| 리눅스 Screen 사용, 명령어 (0) | 2024.02.19 |
|---|---|
| [서버관리] 리눅스 기본 쉘 설정 (0) | 2023.06.30 |
| 알아두기) vcf파일 chr+숫자 position 맞게 정렬 (0) | 2023.06.29 |
| 나의 Matrix Factorization 이야기 (2) (0) | 2023.04.21 |


