| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | ||||||
| 2 | 3 | 4 | 5 | 6 | 7 | 8 |
| 9 | 10 | 11 | 12 | 13 | 14 | 15 |
| 16 | 17 | 18 | 19 | 20 | 21 | 22 |
| 23 | 24 | 25 | 26 | 27 | 28 |
- java
- SWEA
- level1
- WebOS
- level3
- androidstudio
- 안드로이드스튜디오
- 대학원일기
- 프로그래머스
- 컨트리뷰톤
- D3
- 컴퓨터비전
- 대학원
- py
- 파이썬
- build
- Python
- 다시풀기
- MSBuild
- level4
- BFS
- git
- Matrix Factorization
- 휴학
- SQL
- 어렵다
- LEVEL2
- Github
- 자바
- 내휴학생활중의아주큰일
- Today
- Total
bit가 눈 앞에서 왔다갔다
나의 Matrix Factorization 이야기 (2) 본문
교수님께서 결과 나온 matrix 직접 확인해 봤냐고 물어보셨다. drug마다 어떤 건 response가 높을 거고 어떤 건 낮을 테니, drug마다의 상관관계를 봐야 한다고 하셨다.
내 결과를 보고 언니가 보고 충격받아서 drug간의 correlation을 엄청 오래 따져봤었다는 다른 박사과정 선배님한테 찾아가셔서 꽤 오랜 토론을 하셨다. 그 와중에 신입생은 들을수록 새로웠다.
- cell line에 비해 drug는 잘 안나오는게 당연
- 그리고 더 많은 이야기를 하셨는데, 월요일에 정확히 한번 더 물어봐야겠다.
1. column마다 correlation 구하기
가능한 경우들을 대부분 시도해봤다.
다들 고만고만하게 괜찮게 나왔는데 prediction에 대한 것만 상관계수들이 Nan 값이 찍히면서 heat map이 그려지지 않았다.

corr 값들이 어마어마하게 크거나 어마어마하게 작으면 이렇다고 한다.
근데 내 경우는 그렇지 않았고, predicted matrix가 인덱스, 칼럼이 그냥 0,1,2,3 이렇게 바뀌었는데, 인덱스가 겹치지 않아서 Nan값이 가득 채워져 있었다.
그래서 인덱스를 맞춰줬고, 제대로 된 heatmap을 볼 수 있었다.
concate 한 영역 중 23까지가 predict에 해당하니까 제한한다. "이가 없으면 잇몸으로 해야죠."
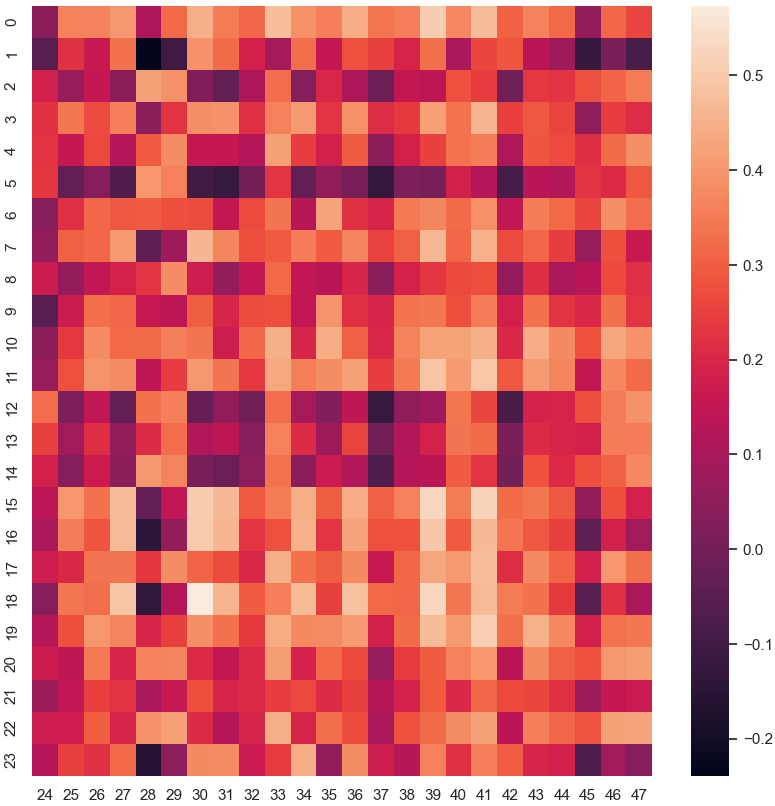
"누가 봐도 빨간데.."
이게 0.9가 나왔다는 게 믿기지가 않았다.
2. PCC..
predict 결과와 target을 csv로 뽑아내서 직접 확인해 봤는데, 아무리 봐도 0.9가 나올 정도가 아니었다. 거의 똑같다는 건데, 누가 봐도 달랐다.
PCC를 하나하나 뜯어봤다.
1) 1차원으로 변환하는 게 일반적이긴 함. (케바케)
np.corrcoef(predicted_matrix2000.values.flatten(),target_matrix2000.values.flatten())[0,1]
그리고 내 경우엔 flatten을 안 해주면 이런 오류가 뜬다.
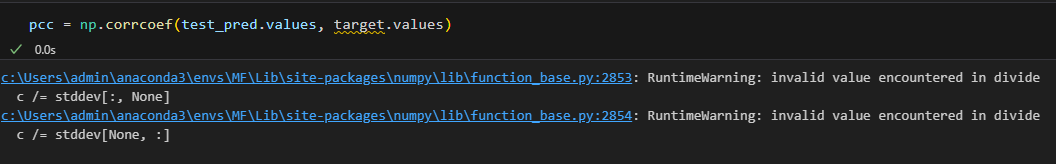
진짜 계속 발생하는 오류라서 너무 괴로웠는데,, flatten을 안 해줘서 생긴 거였다ㅠ
이런 오류는 함수 내부에서 표준편차(stddev)가 0인 경우 0으로 나누는 연산이 발생하면 생기는 문제로
각 row, column의 표준편차가 0이 되어버린다고 하는데, flatten을 하지 않으려면 표준편차가 0이 되지 않도록 먼저 처리해줘야 한다.
(이거 처리 안 해줘도 값이 나오긴 한다.)
-> 이렇게 하면 각 셀마다의 pcc를 구하는 게 아니게 됨. matrix 전체적인 pcc를 구하는 것임.
셀마다 pcc를 구하려면 2차원인 상태로 그대로 둬야 하고, 전체적인걸 구하려면 flatten을 통해 1차원으로 바꿔줘야 한다.
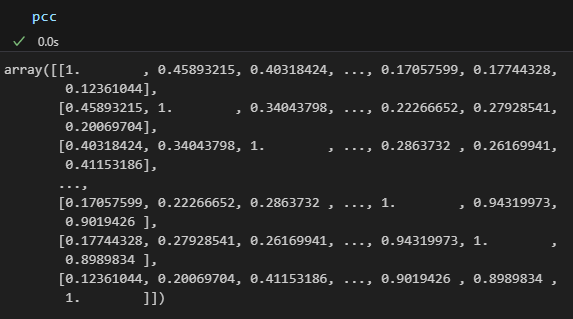
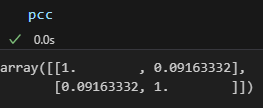
2) 이 상태라면, test_pred와 target의 상관계수 값들의 [0,1]의 위치에 있는 것만 출력한다.
test_pred와 target은 많은 상관계수를 뱉어내고 있는데, flatten() 해주지도 않은 상태에서 [0,1]로 해버리면..
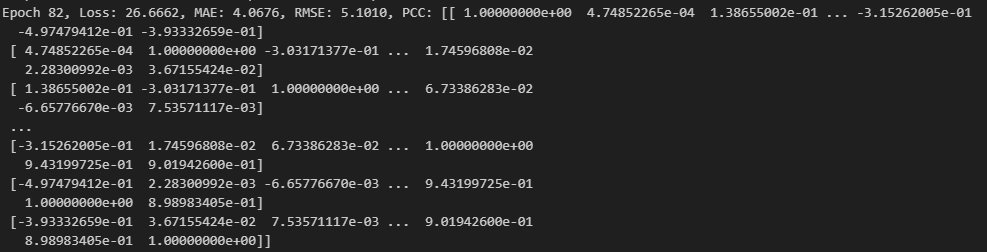
4.7485가 나오는 것^0^...

[0,1]를 붙여준 이유는 flatten 시켜서 matrix 전반에 대한 corr값이 나오게 하고 [0,1] 위치에 있는 matrix 전반적인 값이 나오게 해 주는 거였다.
pearsonr 함수를 사용하려면,

이렇게 하고 [0]을 붙여주는 이유는 pearsonr 함수가

이렇게 결과를 제공하기 때문이었다.
보는 김에 pcc에 대해서도 좀 보게 됐다.

correlation_coeff, p_value = pearsonr(x, y)p_value는 임계값을 정하고 그거보다 작으면 통계적으로 유의미함을 보여주는 수치다.
흔히 사용하는 pcc값은 당연히 correlation_coeff이다.
3. 이제 정상입니다~
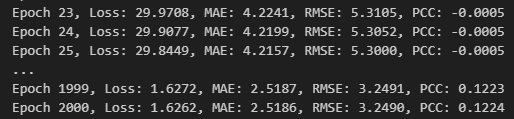
MF는 이렇게 처참하게 나와야 정상이다.
(애초에 코드를 뉴럴네트워크 느낌으로 짜버려서 내가 원하는 목적 달성을 위해서 아예 정상이라고 보기도 어렵겠지만)
언니가 괜찮아요, 충분히 그럴 수 있어요 하시면서 내가 드린 칙촉을 먹으며 벼룩의 간을 떼먹는 거 같네요... 저도 과자 있는데,, 하면서 고마워하셨다...
박사과정 언니한테 상황 말하러 가는 길에 복도에서 여유롭게 바나나 먹으면서 오시는 걸 마주쳤다.
제가 이미 말했어용~ (굿) 하시길래
ㅎ.. 저도 다시 말하러 가요...(굿) 하고 들어갔더니 언니가 (굿)하면서 다 들었어 축하해 그게 공부야~~ 너 빨리 제발 놀아 이제; 5시 반이야.. 퇴근해... 하셨다. 너무 착한 우리 선배님들...
그리고 나는 9시가 넘도록 정리하느라 퇴근하지 못하고 있다.
굿..
이제 월요일에 출근해서 코드를 처음부터 다시 짜면 된다.
'연구' 카테고리의 다른 글
| 리눅스 Screen 사용, 명령어 (0) | 2024.02.19 |
|---|---|
| [서버관리] 리눅스 기본 쉘 설정 (0) | 2023.06.30 |
| 알아두기) vcf파일 chr+숫자 position 맞게 정렬 (0) | 2023.06.29 |
| 나의 Matrix Factorization 이야기 (1) (0) | 2023.04.21 |


